Spesifisitet av deteksjon
I de fleste tilfeller er formålet med primerdesign å maksimere spesifisiteten til PCR.Dette bestemmes av den mer eller mindre forutsigbare påvirkningen av mange variabler.En viktig variabel er sekvensen i 3′enden av primeren.
Viktigere er det mer sannsynlig at PCR-analyser designet for spesifisitet opprettholder høy effektivitet over et bredt dynamisk område, fordi analysen ikke produserer uspesifikke amplifikasjonsprodukter, og dermed konkurrerer med PCR-reagenser eller hemmer hovedamplifikasjonsreaksjonen.
Selvfølgelig er spesifisitet i noen tilfeller ikke det viktigste, for eksempel når målet er å kvantifisere nært beslektede, men forskjellige patogener, kreves det spesielle design-, optimaliserings- og verifikasjonsstandarder.
Smeltekurven er en standardmetode for å vurdere spesifisiteten til amplikoner, i det minste når det gjelder om man skal forsterke et enkelt mål.Det må imidlertid understrekes at smeltekurvene kan være misvisende fordi de for eksempel kan påvirkes av de kombinerte effektene av suboptimale primere og lave malkonsentrasjoner.
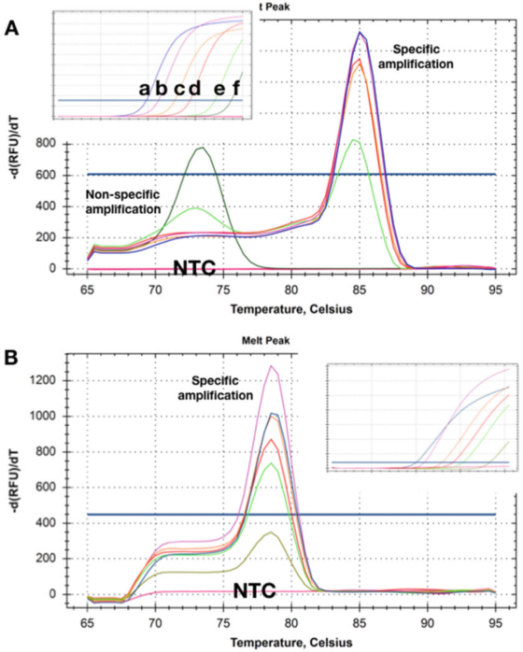
P5 |Smeltekurven viser Tm-skiftene oppnådd fra to deteksjoner av forskjellige mengder av to mål-DNA.
A. Ved høyere konsentrasjoner (ad) er det ingen åpenbar primerdimer etter at qPCR-målingen er fullført.Ettersom malkonsentrasjonen synker til 50 kopier (e), begynner et ikke-spesifikt produkt å vises og blir det eneste produktet med lavest konsentrasjon (f).
B. Testen registrerte samme Tms ved alle målkonsentrasjoner, og det var ingen åpenbar primerdimer selv ved den laveste konsentrasjonen (5 kopier).Ved bruk av disse to deteksjonsmetodene ble det ikke påvist amplifikasjonsprodukter i NTC-er.
P5 viser oppløsningskurvene oppnådd med prøver der malen er tilstede i forskjellige konsentrasjoner.P 5a viser at ved de to laveste konsentrasjonene er Tms for de produserte ikke-spesifikke amplifikasjonsproduktene lavere enn for de spesifikke amplikonene.
Åpenbart kan denne deteksjonsmetoden ikke brukes pålitelig til å oppdage mål som eksisterer i lave konsentrasjoner.
Interessant nok registrerte ikke NTC-er, dvs. prøver uten DNA i det hele tatt, (ikke-spesifikke) amplifikasjonsprodukter, noe som indikerer at genomisk bakgrunns-DNA kan delta i ikke-spesifikk amplifikasjon/polymerisering.
Noen ganger kan slike bakgrunnsprimere og uspesifikk amplifikasjon ikke avhjelpes, men det er ofte mulig å designe en deteksjonsmetode som ikke har uspesifikk amplifikasjon i noen malkonsentrasjon og NTC (P 5b).
Her vil selv registrering av amplifikasjonen av målkonsentrasjonen med en Cq på 35 produsere en spesifikk oppløsningskurve.Tilsvarende viste NTC-er ingen tegn på ikke-spesifikk forsterkning.Noen ganger kan deteksjonsatferden være avhengig av moderluten, og bare ikke-spesifikk amplifikasjon detekteres i visse buffersammensetninger, som kan være relatert til forskjellige Mg2+-konsentrasjoner.
Stabilitet av deteksjon
Optimaliseringen av Ta er et nyttig trinn i den empiriske verifiserings- og optimaliseringsprosessen for qPCR-deteksjon.Den gir en direkte indikasjon på robustheten til primersettet ved å vise temperaturen (eller temperaturområdet) som produserer den laveste Cq uten å forsterke NTC.
Den to til fire gangers forskjellen i sensitivitet er kanskje ikke viktig for personer med høyt mRNA-uttrykk, men for diagnostiske tester kan det bety forskjellen mellom positive og falske negative resultater.
Ta-egenskapene til qPCR-primere kan variere sterkt.Noen tester er lite robuste, og hvis de ikke utføres under den optimale Ta-verdien til primerne, vil de raskt kollapse.
Dette er viktig fordi denne typen påvisning ofte er problematisk i den virkelige verden, og renheten til prøven, konsentrasjonen av DNA eller tilstedeværelsen av annet DNA er kanskje ikke optimal.
I tillegg kan målkopinummeret variere i et stort område, og reagensene, plastredskapene eller instrumentene kan være forskjellige fra de som ble brukt når du setter opp testen.
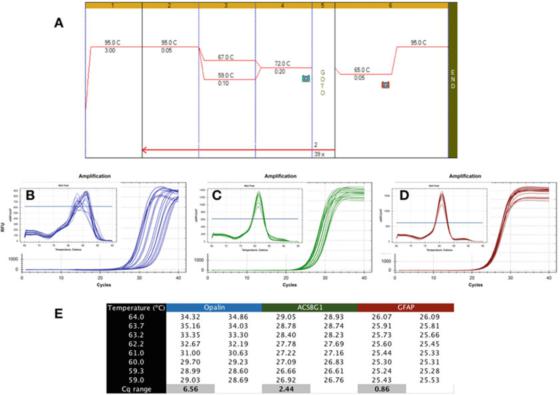
P6|Temperaturgradienten viser ulik robusthet til PCR-deteksjon.
A. Bruk Biolines Sensifast SYBR mastermix (katalognummer BIO-98050) for å utføre PCR på cDNA fremstilt fra human hjerne-RNA.
B. Bruk Bio-Rads CFX qPCR-instrument til å registrere amplifikasjonskartet og oppløsningskurven til apalen (NM_033207, F: GCCATGGAGGAAAGTGACAGACC, R: CTCATGTGTGGGTGATCTCCTAGG).
C. Amplifikasjonsgraf og smeltekurve for ACSBG1 (NM_015162.4, F: CTACACTTCCGGCACCACTGG, R: GTCCACGTGATATTGTCTTGACTCAG).
D. Amplifikasjonsgraf og oppløsningskurve for GFAP (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, R: CGAACCTCCTCCTCGTGGATCTTC).
E. Cqs registrert ved forskjellige glødetemperaturer, som viser forskjellen i Cq registrert under en 7C temperaturgradient.
P 6 viser et typisk resultat av en uønsket test, hvor qPCR ble utført ved bruk av en gradient Tas mellom 59C og 67C (P 6a), ved bruk av primere for tre humane hjernespesifikke gener.
Det kan sees fra amplifikasjonsgrafen at Opalin-primere er langt fra ideelle fordi deres optimale Ta-område er veldig smalt (Figur 6b), det vil si at Cqs er vidt spredt, noe som resulterer i at Cqs blir betydelig sammenlignet med deres optimale Cqs Low.
Denne deteksjonsmetoden er ustabil og kan føre til suboptimal forsterkning.Derfor bør dette primerparet redesignes.I tillegg viser smeltekurveanalysen (innfelt) at spesifisiteten til denne deteksjonsmetoden også kan være problematisk, fordi smeltekurven til hver Ta er forskjellig.
ACSBG1-deteksjonsmetoden vist i P 6c er mer robust enn Opalin-deteksjonsmetoden ovenfor, men den er fortsatt langt fra ideell, og det er sannsynlig at den kan forbedres.
Vi understreker imidlertid at det ikke er noen nødvendig sammenheng mellom robusthet og spesifisitet, fordi oppløsningskurven produsert ved denne deteksjonsmetoden viser samme toppverdi i alle Tas (innfelt).
På den annen side er robusthetstesten mye mer tolerant, og produserer lignende Cq-er i et bredt spekter av Tas, som i GFAP-testen vist i P 6d.
Forskjellen i Cqs oppnådd i samme 8 grader Celsius-område er mindre enn 1, og oppløsningskurven (innsatt) bekrefter deteksjonsegenskapene i dette temperaturområdet.Det er verdt å merke seg at den beregnede Tas og det faktiske Ta-området kan være svært forskjellige.
Det er mange retningslinjer designet for å hjelpe forskere med å designe effektive primere, de fleste er basert på lenge etablerte regler og mye oppmerksomhet har blitt viet til 3′enden av primerne.Det anbefales ofte å inkludere en G eller C i 3'-enden og to G- eller C-baser (GC-klemme), men ikke mer enn to av de siste 5 basene.
I praksis kan disse reglene veilede forskere, men de er ikke nødvendigvis riktige under alle omstendigheter.
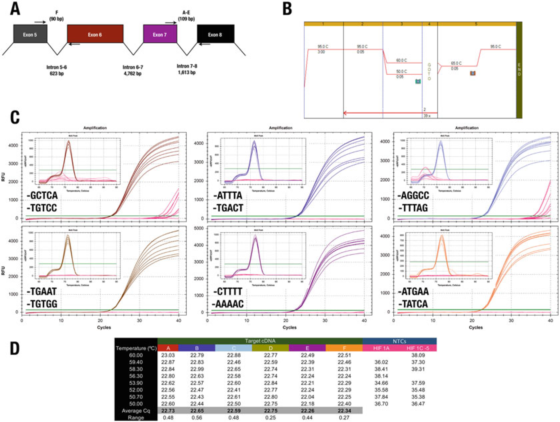
P7 |3′enden av primeren har liten effekt på spesifisitet eller effektivitet.
A. Posisjonen til primerne for det humane HIF-1α (NM_181054.2) genet.
B. Bruk Agilent Brilliant III SYBR Grønn moderlut (kat.nr. 600882) for å forsterke seks testelementer.
C. Amplifikasjonsgraf og smeltekurve registrert av Bio-Rads CFX qPCR instrument og 3'ende primere.NTC-er vises i rødt.
D. Cqs registrering av hvert testelement
For eksempel motsier resultatet i P 7 3′enderegelen.Alle design gir stort sett de samme resultatene, med bare to primerkombinasjoner som fører til ikke-spesifikk amplifikasjon i NTC.
Vi kan imidlertid ikke støtte effekten av GC-klippet, fordi bruk av A eller T som maksimalt 30 baser i dette tilfellet ikke reduserer spesifisiteten.
Test C, der F-primeren slutter på GGCC, registrerte Cqs i NTC-er, noe som indikerer at man kanskje vil unngå disse sekvensene i 30-enden.Vi understreker at den eneste måten å bestemme den beste 3'ende-sekvensen til et primerpar er å evaluere noen kandidatprimere eksperimentelt.
Forsterkningseffektivitet
Viktigere, selv om ikke-spesifikk PCR-deteksjon aldri kan bli spesifikk, kan amplifikasjonseffektiviteten justeres og maksimeres på mange forskjellige måter ved å endre enzymet, moderluten, tilsetningsstoffene og syklusforholdene.
For å evaluere effektiviteten av PCR-deteksjon er det best å bruke en seriefortynning på 10 eller 5 ganger målnukleinsyren, det vil si "standardkurvemetoden".
Hvis PCR-amplikoner eller syntetiske DNA-mål brukes til å generere en standardkurve, bør seriefortynninger av disse målene blandes med en konstant mengde bakgrunns-DNA (som genomisk DNA).
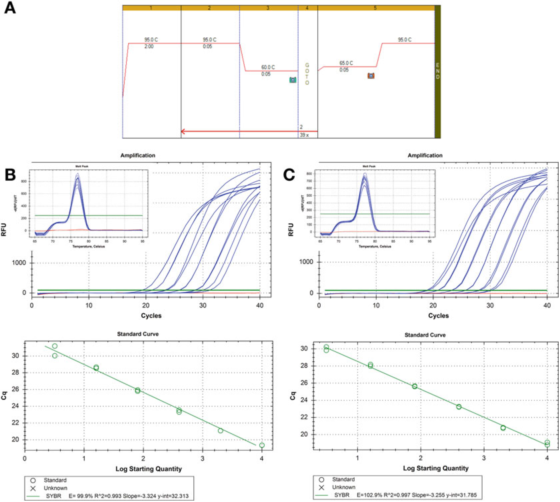
P8 |Fortynningskurve for å evaluere effektiviteten til PCR.
A. Bruk primere for HIF-1: F: AAGAACTTTTAGGCCGCTCA og R: TGTCCTGTGGTGACTTGTCC og Agilent's Brilliant III SYBR Green mastermix (katalognummer 600882) for PCR- og smeltekurveforhold.
B. 100 ng RNA ble omvendt transkribert, fortynnet 2 ganger, og serielt fortynnede cDNA-prøver ble fortynnet 5 ganger til 1 ng humant genomisk DNA.Smeltekurven er vist i innlegget.
C. RT-reaksjonen, fortynningen og seriefortynningen ble gjentatt for den andre cDNA-prøven, og resultatene var like.
P 8 viser to standardkurver, ved bruk av samme deteksjonsmetode på to forskjellige cDNA-prøver, resultatet er samme effektivitet, ca. 100 %, og R2-verdien er også lik, det vil si graden av tilpasning mellom de eksperimentelle dataene og regresjonslinjen eller dataene Grad av linearitet.
De to standardkurvene er sammenlignbare, men ikke helt like.Hvis formålet er å kvantifisere målet nøyaktig, må det bemerkes at det er uakseptabelt å gi en kopiantallsberegning uten å forklare usikkerheten
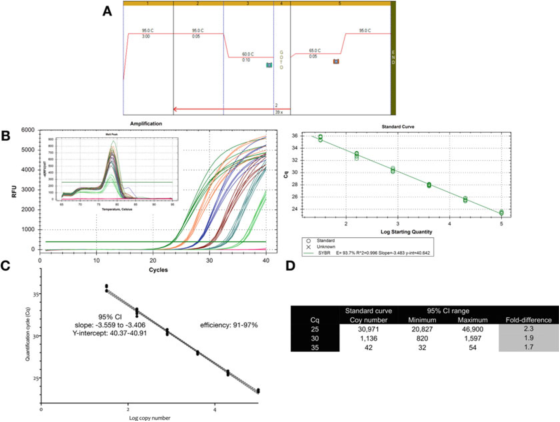
P9 |Måleusikkerhet knyttet til kvantifisering ved bruk av standardkurve.
A. Bruk primere for GAPDH (NM_002046) for å utføre PCR og smeltekurveforhold.F: ACAGTTGCCATGTAGACC og R: TAACTGGTTGAGCACAGG og Biolines Sensifast SYBR mastermix (katalognummer BIO-98050).
B. Amplifikasjonsdiagram, smeltekurve og standardkurve registrert med Bio-Rads CFX qPCR-instrument.
C. Standard kurvegraf og 95 % konfidensintervall (CI).
D. Kopitallet og 95 % konfidensintervall for de tre Cq-verdiene utledet fra fortynningskurven.
P 9 viser at for en optimalisert test er den iboende variasjonen til en enkelt standardkurve omtrent 2 ganger (95 % konfidensintervall, minimum til maksimum), som kan være den minste variasjonen som kan forventes.
Relatert produkt:
Innleggstid: 30. september 2021