Utgangsmateriale: RNA
Kvantitativ revers transkripsjon PCR (RT-qPCR) er en eksperimentell metode som brukes i PCR-eksperimenter med RNA som utgangsmateriale.I denne metoden blir total-RNA eller messenger-RNA (mRNA) først transkribert til komplementært DNA (cDNA) ved revers transkriptase.Deretter ble en qPCR-reaksjon utført ved bruk av cDNA som en mal.RT-qPCR har blitt brukt i en rekke molekylærbiologiske applikasjoner, inkludert genekspresjonsanalyse, RNA-interferensvalidering, mikroarray-validering, patogendeteksjon, genetisk testing og sykdomsforskning.
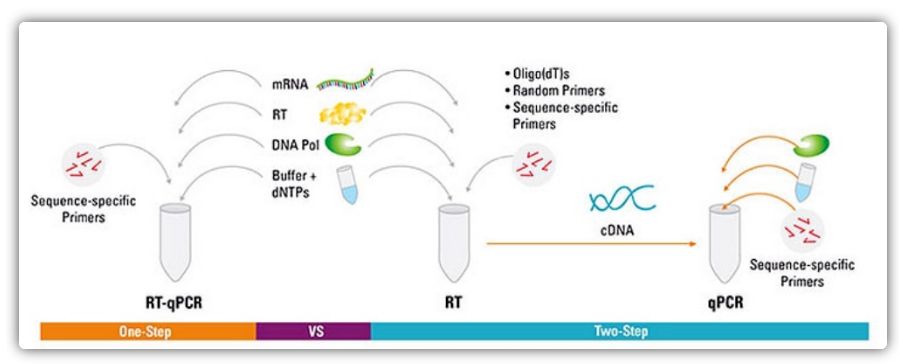
Ett-trinns og to-trinns metoder for RT-qPCR
RT-qPCR kan oppnås ved en ett- eller to-trinns metode.Ett-trinns RT-qPCR kombinerer revers transkripsjon og PCR-amplifikasjon, slik at revers transkriptase og DNA-polymerase fullfører reaksjonen i samme rør under de samme bufferforholdene.Ett-trinns RT-qPCR krever kun bruk av sekvensspesifikke primere.I to-trinns RT-qPCR utføres revers transkripsjon og PCR-amplifikasjon i to rør, ved bruk av forskjellige optimaliserte buffere, reaksjonsbetingelser og primerdesignstrategier.
| Fordel | Ulempe | |
| Et skritt | Denne metoden har mindre eksperimentell feil da begge reaksjonene gjøres i ett rør
Færre pipetteringstrinn reduserer risikoen for kontaminering
Egnet for forsterkning/screening med høy gjennomstrømning, rask og reproduserbar | To-trinns reaksjoner kan ikke optimaliseres separat
Siden reaksjonsbetingelsene kompromitteres ved å kombinere totrinnsreaksjonen, er ikke følsomheten like god som for totrinnsmetoden
Antall mål oppdaget av en enkelt prøve er lite |
| To trinn | Evne til å lage stabile cDNA-biblioteker som kan lagres i lange perioder og brukes i flere reaksjoner
Målgener og referansegener kan amplifiseres fra samme cDNA-bibliotek uten behov for flere cDNA-biblioteker
Reaksjonsbuffere og reaksjonsbetingelser som muliggjør optimalisering av enkeltreaksjonskjøringer
Fleksibelt valg av triggerbetingelser | Bruk av flere rør og flere pipetteringstrinn øker risikoen for DNA-kontaminering, og tidkrevende.
Krever mer optimalisering enn ett-trinnsmetoden |
Relaterte produkter:
RT-qPCR Easyᵀᴹ (One Step)-SYBR Grønn I
RT-qPCR Easyᵀᴹ (ett trinn)-Taqman
RT Easyᵀᴹ I Master Premix for første-strengs CDNA-syntese
Real Time PCR Easyᵀᴹ-SYBR Green I Kit
Seleksjon av totalt RNA og mRNA
Når du designer et RT-qPCR-eksperiment, er det viktig å bestemme om du skal bruke totalt RNA eller renset mRNA som mal for revers transkripsjon.Selv om mRNA kan være i stand til å gi litt høyere sensitivitet, brukes fortsatt totalt RNA ofte.Grunnen til dette er at total-RNA har en viktigere fordel som utgangsmateriale enn mRNA.For det første krever prosessen færre rensetrinn, noe som sikrer bedre kvantitativ gjenvinning av mal og bedre normalisering av resultater til startcelletall.For det andre unngår den mRNA-anrikningstrinnet, som kan unngå muligheten for skjeve resultater på grunn av forskjellige gjenvinninger av forskjellige mRNA.Totalt sett, siden i de fleste applikasjoner er den relative kvantifiseringen av målgenet viktigere enn den absolutte sensitiviteten til deteksjonen, er total RNA mer egnet i de fleste tilfeller.
Revers transkripsjonsprimer
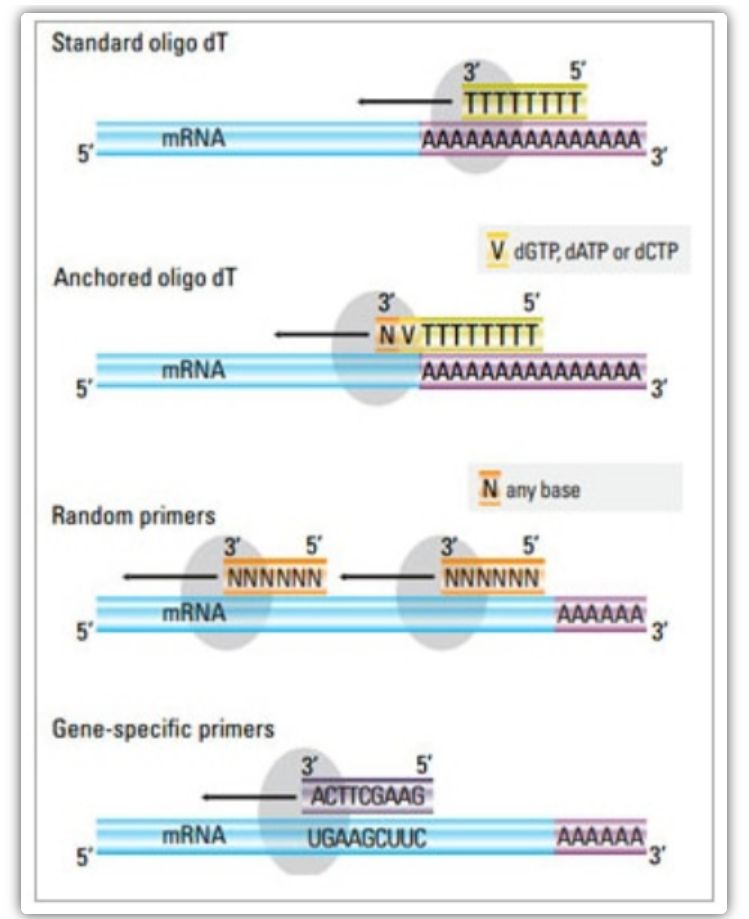
I to-trinnsmetoden kan tre forskjellige metoder brukes for å prime cDNA-reaksjonen: oligo(dT)-primere, tilfeldige primere eller sekvensspesifikke primere.Vanligvis brukes oligo(dT)-primere og tilfeldige primere i kombinasjon.Disse primerne annealer til mal-mRNA-strengen og gir revers transkriptase med et utgangspunkt for syntese.
| Primer valg | Struktur og funksjon | Fordel | Ulempe |
| Oligo(dT) primer (eller forankret oligo(dT) primer) | Forlenget annealing til tyminrester ved poly(A)-halen til mRNA;ankeroligo(dT)-primer inneholder en G, C eller A i 3'-enden (ankersted) | Syntese av full-lengde cDNA fra poly(A)-hale-mRNA
Gjelder når mindre utgangsmateriale er tilgjengelig
Forankringssted sikrer at oligo(dT)-primeren binder seg til 5′ poly(A) halen av mRNA. | Kun egnet for amplifisering av gener med poly(A)-haler
Skaff cDNA avkortet fra primingsstedet*2 i poly(A)
Biased for å binde til 3′-enden*
*Denne muligheten minimeres hvis forankrede oligo(dT)-primere brukes |
| tilfeldig primer
| 6 til 9 baser i lengde, som kan anneale til flere steder under RNA-transkripsjon | Anneal til alle RNA (tRNA, rRNA og mRNA)
Egnet for transkripsjoner med betydelig sekundær struktur, eller når mindre utgangsmateriale er tilgjengelig
Høyt cDNA-utbytte | cDNA er omvendt transkribert fra alt RNA, som vanligvis ikke er ønsket og kan fortynne signalet til mål-mRNA
få avkortet cDNA |
| sekvensspesifikke primere | Tilpassede primere rettet mot spesifikke mRNA-sekvenser | spesifikt cDNA-bibliotek
Forbedre følsomheten
Bruke omvendte qPCR-primere | Bare begrenset til syntesen av et enkelt målgen |
Revers transkriptase
Revers transkriptase er et enzym som bruker RNA til å syntetisere DNA.Noen revers transkriptaser har RNase-aktivitet og kan bryte ned RNA-tråder i RNA-DNA-hybridtråder etter transkripsjon.Hvis den ikke har RNase-enzymatisk aktivitet, kan RNaseH tilsettes for høyere qPCR-effektivitet.Vanlig brukte enzymer inkluderer Moloney murine leukemivirus revers transkriptase og fuglemyeloblastomvirus revers transkriptase.For RT-qPCR er det ideelt å velge en revers transkriptase med høyere termostabilitet, slik at cDNA-syntese kan utføres ved høyere temperaturer, noe som sikrer vellykket transkripsjon av RNA-er med høyere sekundærstruktur, samtidig som de opprettholder deres fulle aktivitet gjennom hele reaksjonen, noe som resulterer i høyere cDNA-utbytter.
Relaterte produkter:
Foreasy M-MLV revers transkriptase
RNase H-aktivitet av revers transkriptase
RNaseH er i stand til å bryte ned RNA-tråder fra RNA-DNA-duplekser, noe som tillater effektiv syntese av dobbelttrådet DNA.Imidlertid, når du bruker lang mRNA som en mal, kan RNA bli degradert for tidlig, noe som resulterer i avkortet cDNA.Derfor er det ofte fordelaktig å minimere RNaseH-aktivitet under cDNA-kloning hvis syntese av lange transkripsjoner er ønsket.I motsetning til dette er revers transkriptaser med RNase H-aktivitet ofte fordelaktige for qPCR-applikasjoner fordi de øker smeltingen av RNA-DNA-duplekser under den første PCR-syklusen.
Primer design
PCR-primere brukt for qPCR-trinnet i RT-qPCR bør ideelt sett utformes for å spenne over et ekson-ekson-kryss, der en amplifikasjonsprimer potensielt kan spenne over en faktisk ekson-intron-grense.Siden intronholdige genomiske DNA-sekvenser ikke er amplifisert, reduserer denne utformingen risikoen for falske positiver amplifisert fra kontaminerende genomisk DNA.
Hvis primere ikke kan utformes for å skille eksoner eller ekson-ekson-grenser, kan det være nødvendig å behandle RNA-prøver med RNase-fri DNase I eller dsDNase for å fjerne genomisk DNA-kontaminering.
RT-qPCR-kontroll
En negativ transkripsjonskontroll (-RT-kontroll) bør inkluderes i alle RT-qPCR-eksperimenter for å oppdage DNA-kontaminering (som genomisk DNA eller PCR-produkter fra tidligere reaksjoner).Denne kontrollen inneholder alle reaksjonskomponenter unntatt revers transkriptase.Siden revers transkripsjon ikke forekommer med denne kontrollen, hvis PCR-amplifikasjon observeres, er kontaminering fra DNA mest sannsynlig.
Innleggstid: Aug-02-2022








